模擬5億年自然演化史,全新蛋白質大模型ESM3誕生!前Meta老將力作LeCun轉贊

繼AlphaFold 3更新後,我們又看到了一個生命科學領域的大模型ESM3。
模型開發團隊來自於一家名為Evolutionary Scale AI的新創公司,團隊負責人Alex Rives在推特上宣布了模型發布的消息。

這個令人振奮的消息也得到了Yann LeCun的轉發,他表示,你們這個公司有點「悶聲發大財」的意思。

相比AlphaFold系列,ESM3有什麼競爭優勢?
首先就是Meta團隊輕車熟路的-開源。

雖然模型API仍處於內測階段,需要申請試用資格,但模型程式碼已經放到了GitHub上。而且公司也會與AWS和英偉達雲端運算平台合作,方便開發者使用和部署。
倉庫地址:https://github.com/evolutionaryscale/esm
但比較遺憾的是,目前在HuggingFace倉庫中還沒有公開模型權重。英偉達官方部落格顯示,ESM3將在Nvidia BieNeMo平台提供一個小型開源版本的程式碼和權重,但僅限於非商業用途。
倉庫位址:https://huggingface.co/EvolutionaryScale/esm3-sm-open-v1/tree/main
此外,ESM3與不同於模擬多種生物分子的AlphaFold 3,只專注於蛋白質,但可以同時推理其序列、結構和功能,這種多模態能力屬於領域首創。
更讓人耳目一新的是,ESM3在自然界中27.8億個多樣化蛋白質上進行訓練,逐漸學習到了演化過程如何讓蛋白質改變。
從這個角度來看,ESM的推理過程可以被視為「演化模擬器」,這為目前的生命科學研究開啟了全新的視野。團隊甚至在官網文章中提出了「模擬5億年進化」的標語。
或許你已經注意到了,ESM這個名字和Meta之前的蛋白質模型ESMFold非常相似。
這並不是有意擦邊。事實上,Evolutionary Scale這家新創公司就是Meta-FAIR蛋白質小組的前成員創辦的,而公司的首席科學家Alex Rives正是這個已解散團隊的前負責人。

去年8月,在Meta的「效率年」中,祖克柏選擇解散了只有十幾名科學家的蛋白質小組,讓公司專注於更有獲利前景的研究。

但Rives並沒有被Meta的這種舉動嚇倒,而是決定自立門戶,他們目前已經籌集了1.42億美元的種子資金。
那麼就來仔細看看,這次的ESM3具體有哪些新內容呢?

ESM3:生物學的前沿語言模型
生命科學並不像我們想像的那樣神秘莫測、不可捉摸。
蛋白質分子雖然有難以置信的多樣性和動態變化,但是它的合成遵循嚴密的演算法與流程。如果把它看成一門技術,其先進程度遠遠超過任何人類創造的工程。
生物學,就是厚厚的密碼本。
只不過,這個密碼本來是用我們尚未理解的語言寫就的,即使是當今最強超算上運行的工具也不過觸及皮毛。
如果人類能夠閱讀,甚至是書寫「生命代碼」,就能使生物學變得可程式化。試誤法將被邏輯取代,費力的實驗將被模擬取代。
ESM3就是朝這個宏偉願景邁出的一步,是迄今為止首個能同時對蛋白質的序列、結構和功能進行推理的生成模型。
過去五年LLM的突飛猛進,也讓ESM團隊發現了Scaling Law的威力,他們發現,同樣的模式也適用於生物學。
隨著訓練資料以及參數規模的擴大,模型會加深對生物學基本原理的理解,並能更好地預測、設計生物結構和功能。
因此,ESM3的開發想法也與Scaling Law一脈相承,其規模比上一代ESM大大擴展,資料量提高了60倍,訓練計算量提高了25倍,並且是具有原生多模態的生成模型。
ESM3的訓練過程囊括了地球自然環境的多樣性——數十億種蛋白質,從亞馬遜雨林到海洋深處,小到土壤中的微生物,極端到深海熱泉。
HuggingFace上的模型卡顯示,訓練集中天然蛋白質數量達到27.8億,並透過合成資料增強到31.5億個序列、2.36個結構以及5.39億個帶有功能註釋的蛋白質,token總數達到7710B。
模型訓練參數總量達到98B,使用了超過10 24 FLOPS的算力。團隊似乎與英偉達緊密合作,訓練使用了Andromeda集群,是當今吞吐量最高的GPU集群之一,部署了最先進的H100 GPU和Quantum-2 InfiniBand網路。

網頁來源:https://andromeda.ai/
他們表示「相信ESM3的計算總量是有史以來生物模型之最」。
推理蛋白質的序列、結構與功能
處理文本的語言模型一般以token為基本單位,但多模態的蛋白質模型較為複雜,需要將序列、三維結構和功能都轉換為離散的字母來表示。
為了更好地擴展訓練規模、釋放模型的「湧現」生成潛力,ESM3使用的詞彙在同一語言模型中能夠很好地連接序列、結構和功能,進行聯合推理。
有別於GPT等語言模型,ESM3的訓練目標繼承於遮罩語言模型(masked language modeling objective)。
每個蛋白質的序列、結構、和功能的部分位置會被掩碼,模型在訓練過程中需要逐漸理解三者之間的深層聯繫,從而預測掩碼位置。如果遮蔽所有位置的標記,就相當於執行生成任務。
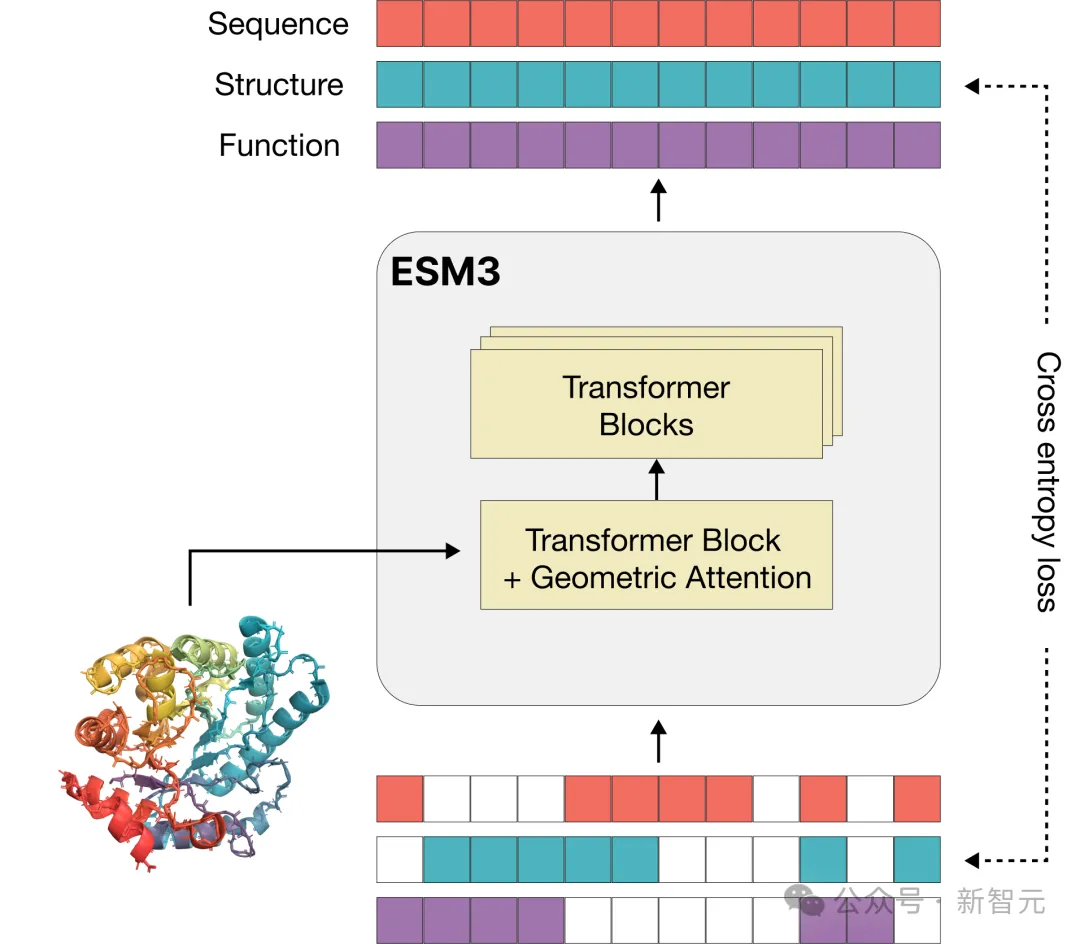
由於在蛋白質的序列、結構和功能上聯合訓練,對這三種模態可以任意進行掩碼和預測,因此ESM3實現了「全對全」預測或生成(all to all)。
也就是說,模型的輸入可以是部分或完全指定的三種模態的任意組合。這種強大的多模態推理能力有很強的應用價值,科學家能以前所未有的彈性和控制設計出全新的蛋白質。
例如,可以提示模型結合結構、序列和功能,提出PET酶活性位點的潛在支架結構。 PET是一種常用的塑料,如果PET酶設計成功,就能用於高效分解塑膠廢物。

ESM3透過序列、結構和功能的多模態提示設計PET酶活性位點的支架
Evolutionary Scale的聯合創始人兼兼工程副總裁Tom Sercu表示,在內部測試中,ESM在應對各種複雜提示時展現了令人印象深刻的創造力。
「它能夠解決一個極其困難的蛋白質設計問題,創造一種新型綠色螢光蛋白。ESM3能夠幫助科學家加速工作,開闢新的可能性——我們期待看到它在未來對生命科學研究的貢獻。」
當數十億個蛋白質來自演化時間軸上的不同位置,具有豐富的多樣性時,模型還能學到模擬演化的能力。
能力隨規模湧現
正如LLM在規模擴展中「湧現」出了語言理解、推理等能力,在解決有挑戰性的蛋白質設計任務時,ESM3也隨規模增加逐漸顯現能力,其中一個重要的能力就是原子級協調。
例如,提示中可能指定組成蛋白質的兩個胺基酸需要在序列位置上相近,但在結構中相距較遠。這衡量了模型在結構生成任務中達到原子級精度的能力。
這對於設計功能性蛋白質至關重要,而ESM3解決這類複雜生成任務的能力可以隨著規模增加而逐漸提高。
不僅如此,在訓練完成後,ESM3的能力還有進一步提升的空間,其機制類似LLM常用的RLHF方法。
但差異在於,ESM3並不是從人類接受回饋,而是可以評估自身的生成質量,進行自我改進,也可以結合現有的實驗數據和濕實驗結果,讓ESM3的生成與生物學結果保持對齊。
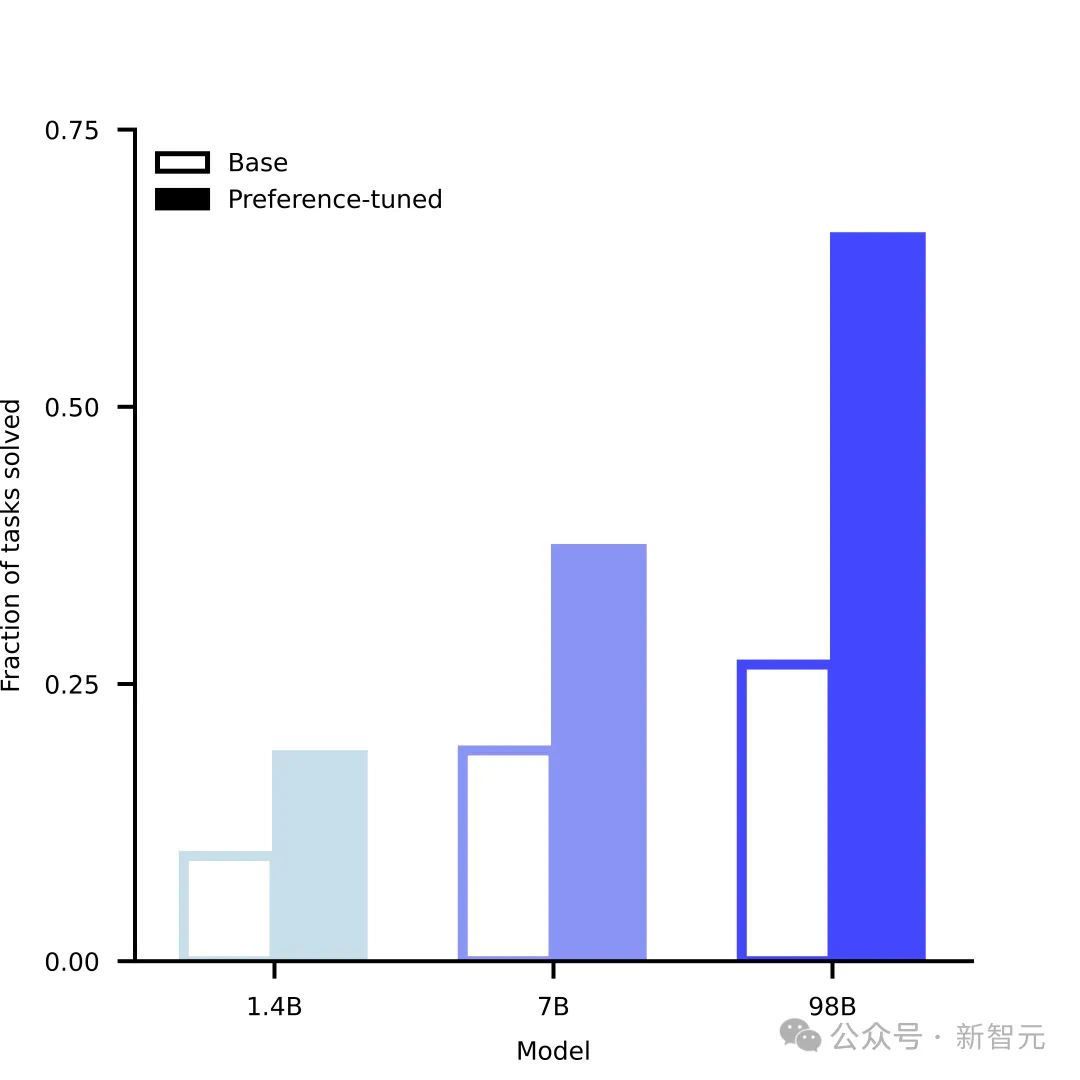
模型生成能力隨規模成長,且微調的提升效果明顯
模擬5億年的演化
在發表的論文中,ESM3團隊詳細介紹了他們在模型上觀察到的「模擬演化」功能。

論文地址:https://evolutionaryscale-public.s3.us-east-2.amazonaws.com/research/esm3.pdf
綠色螢光蛋白(Green Fluorescent Protein,GFP)及其螢光蛋白家族是自然界中最美麗的蛋白質之一,但它們只存在於「生命之樹」的幾個分支中。
但GFP不僅僅是美麗而已,它包含一種螢光髮色團(fluorescent chromophore)。這種分子可以吸收短波長的單色光子、捕獲部分能量,再釋放出波長較長的另一種單色光子。例如,自然界存在的GFP可以吸收藍光並發出綠光。
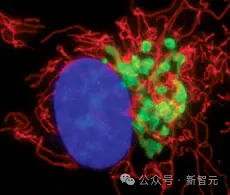
由於這種特性,GFP能夠作為標記,幫助科學家在細胞內觀察蛋白質,成為了生物學中最廣泛使用的工具之一,GFP的發現也因此獲得了諾貝爾獎。
GFP的功能如此獨特且有用,結構也非常罕見:由十一條鏈組成的桶狀結構,中間穿過一條螺旋。折疊後進行自發性反應,在蛋白質中心的分子會重新排列,產生螢光髮色團。
這種機制是獨一無二的。沒有其他已知的蛋白質能夠自發地從其自身結構中形成螢光髮色團,這表明即使在自然界中,產生螢光也是相當罕見且困難的。
為了在實驗室有更廣泛的應用,科學家嘗試加入突變或改變顏色,進行人工合成。最新的機器學習技術能夠搜尋到序列差異高達20%的變體,但功能性GFP的主要來源依舊是自然界而非蛋白質工程。
想在自然界中找到更多的變體並不簡單,因為新螢光蛋白的進化需要漫長的時間——GFP所屬家族的歷史相當久遠,它們從祖先序列中分化出來的時間點能追溯到數億年前。
這個棘手的問題或許能在ESM3這裡解決。
將天然GFP核心結構中幾個位點的資訊作為提示,並使用CoT技巧,ESM3成功產生了新型GFP的候選者。
這種生成絕不可能是隨機撞大運或全域搜索,因為可能的序列和結構的組合起來會達到天文數字——20 229 x 4096 229 ,比可見宇宙中所有的原子數加起來還要多。
在首次實驗中,團隊測試了ESM3產生的96個候選蛋白,其中出現了成功的發光樣本,而且存在一種十分獨特的結構,與自然界中任何蛋白質都相差甚遠。
在另一組96個候選樣本中,發現了幾種亮度與天然GFP相似的蛋白質,其中一種亮度最高的蛋白質被命名為esmGFP,與最接近的天然螢光蛋白相比有96個突變(在229個胺基酸組成的序列中,有58%的相似部分)。
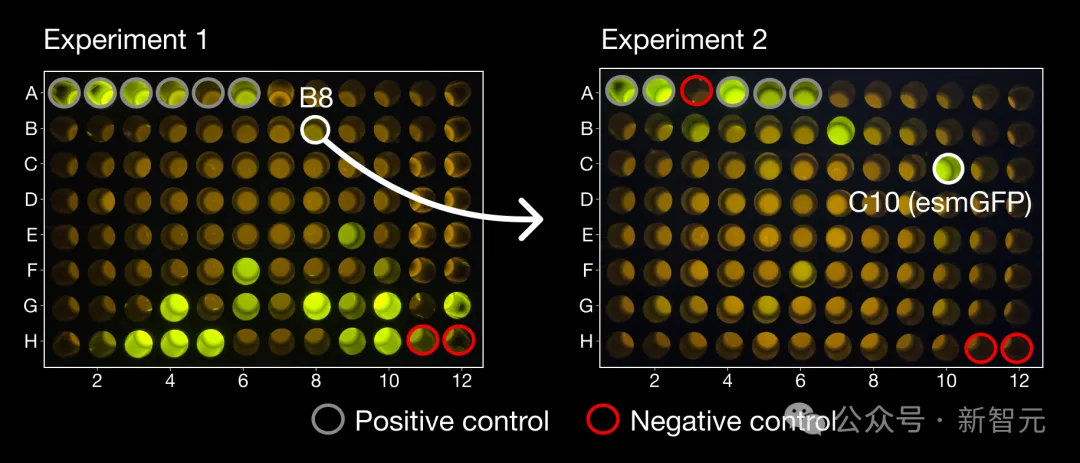
左側是與所有已知GFP都相差較大的生成物B8,從B8開始,ESM3生成了右圖的esmGFP
與自然演化不同,蛋白質語言模型並不在演化約束內明確運作。
但為了讓ESM3解決其預測下一個掩碼token的訓練任務,模型必須學習演化如何在潛在蛋白質空間中演變。
從這個意義上來說,ESM3生成與天然蛋白十分相似的esmGFP的過程,可以被視為演化模擬器。
對esmGFP進行傳統的進化分析是自相矛盾的,因為它是在自然過程之外創造的,但仍可以從進化生物學的工具中獲得洞見,了解一個蛋白質通過自然進化與其最近的序列鄰居分化所需的時間。
因此,研究團隊使用演化生物學的方法,把esmGFP當成自然界新發現的蛋白質來分析。他們估計,esmGFP等效於演化模擬器執行的超過5 億年的自然演化。

esmGFP的渲染圖
開放模型
自成立以來,ESM計畫(ESM project)一直致力於透過發布程式碼和模型來實現開放科學。目前仍能在GitHub和HuggingFace上找到團隊在幾年前發布的程式碼和模型權重。
倉庫位址:https://huggingface.co/facebook/esm2_t36_3B_UR50D/tree/main
看到ESM模型在研究和工業領域的創造性和有影響力的應用,可謂是令人驚嘆:
- Hie等人使用ESM-1v和ESM-1b來演化抗體,改善了結合親和力、熱穩定性和病毒中和等治療相關特性。
- BioNTech和InstaDeep微調了一個ESM語言模型,用於檢測COVID刺突蛋白中的變異,成功地在WHO指定之前標記了所有16種關注變異。
- Brandes等人使用ESM-1b來預測突變的臨床效果,目前仍是完成該重要任務的最強方法。
- Marsiglia等人使用ESM-1v來設計新的抗CRISPR蛋白變體,這些變體在保持目標編輯功能的同時,減少了對非目標副作用。
- Shanker等人使用ESM-IF1引導多樣蛋白的演化,包括實驗室驗證的對SARS-CoV-2高效抗體。
- Yu等人微調了ESM-1b來預測酶的功能,包括稀有和研究不足的酶,並通過實驗驗證了預測結果。
- Rosen等人使用ESM2嵌入來建構單細胞基礎模型中的基因表示。
- Høie等人微調了ESM-IF1在抗體結構上的表現,在CDR區域的序列恢復中達到了最先進的性能,設計出了高結合親和力的抗體。
而這些,只是建立在ESM平台上的驚人工作的一小部分!
如今,團隊正式宣布,將發布一個ESM3 1.4B參數版本的權重和程式碼,以便科學家和開發人員能夠基於ESM3的理念和架構進行建構。