超越AF2?Iambic、英偉達、加州理工學院開發多尺度深度生成模型,進行狀態特異性蛋白質-配體複合物結構預測

由蛋白質和小分子配體形成的結合複合物無所不在,對生命至關重要。雖然最近科學家在蛋白質結構預測方面取得了進展,但現有演算法無法系統地預測結合配體結構及其對蛋白質折疊的調節作用。
為了解決這種差異,AI 製藥公司Iambic Therapeutics、英偉達(Nvidia Corporation)以及加州理工學院(California Institute of Technology)的研究人員提出了NeuralPLexer,這是一種計算方法,可以只使用蛋白質序列和配體分子圖輸入直接預測蛋白質-配體複合物結構。
NeuralPLexer 採用深度生成模型以原子分辨率對結合複合物的三維結構及其構象變化進行取樣。該模型基於擴散過程,該過程結合了基本的生物物理約束和多尺度幾何深度學習系統,以分層方式迭代採樣殘留級接觸圖和所有重原子座標。
NeuralPLexer 預測與酶工程和藥物發現中重要靶點的結構測定實驗相一致,其在蛋白質組規模上加速功能蛋白和小分子設計方面擁有巨大潛力。
研究以「State-specific protein–ligand complex structure prediction with a multiscale deep generative model」為題,於2024 年2 月12 日發佈在《Nature Machine Intelligence》。

靜態蛋白質結構預測不足以支持藥物設計
深度學習在從一維胺基酸序列預測蛋白質結構方面取得了巨大進展。最先進的蛋白質結構預測網絡,例如AlphaFold2 (AF2),採用基於蛋白質結構的進化、物理和幾何約束的預測管線。具體來說,從多重序列比對(MSA)或蛋白質語言模型(PLM)和專門的神經網路中提取的進化約束,與基於序列的資訊和幾何表示系統地結合,從而實現端到端的三維(3D )結構預測。
雖然在預測蛋白質靜態結構方面取得了巨大成功,但蛋白質折疊問題的這種單一結構公式提供了有關蛋白質功能的不完整信息,並且還被發現不足以用於基於結構的藥物設計。
生成式深度學習是一種替代範式
然而,與受體構象的實質變化相結合的蛋白質-配體複合物的計算模型,受到模擬緩慢蛋白質狀態轉變的高昂成本的阻礙。生成式深度學習的最新發展提供了一種替代範式,並且在理解複雜視覺和語言領域方面取得了實質進展。
生成建模的兩個值得注意的策略包括(1)自回歸模型,在序列資料(例如自然語言和基因組學)的Transformer 網路中廣泛採用,基於順序過程;(2) 基於擴散的生成模型,利用隨機過程透過從先驗分佈中取樣並使用神經網路逐步逆轉噪音過程來產生資料。
科學家已經證明,深度生成模型能夠產生具有經過實驗驗證的功能的從頭設計的蛋白質,包括用於蛋白質序列設計的語言模型和用於蛋白質主鏈生成的擴散模型。擴散模型可以有效地模擬蛋白質骨架以外的分子結構,特別是在分子對接和基於結構的藥物設計方面。
然而,目前為止,還沒有團隊開發出能夠以原子分辨率直接預測結合複雜結構且精度可與結構測定實驗相媲美的生成模型。
深度生成模型預測蛋白質-配體複雜結構
在最新的研究中,Iambic、英偉達、加州理工學院團隊介紹了NeuralPLexer,這是一種計算系統,它使用由生物物理歸納偏差提供的深度生成模型來預測蛋白質-配體複雜結構。此方法可以以從PLM 獲得的輔助特徵和從實驗解析的同源物或計算模型檢索的模板蛋白質結構為條件,直接產生給定蛋白質序列和配體分子圖輸入的結合複合物的結構集合。
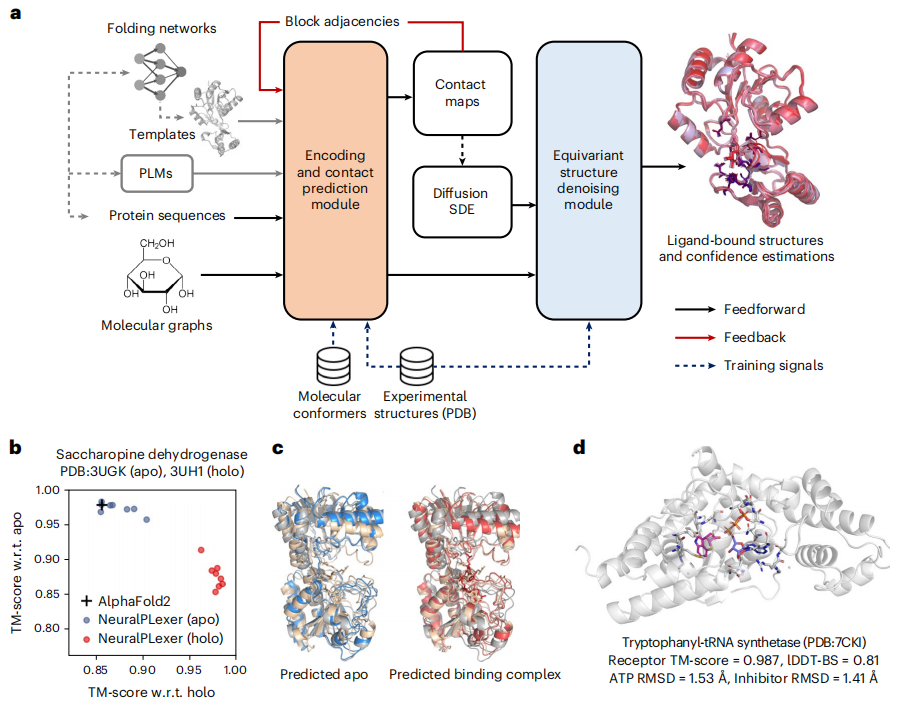
圖示:NeuralPLexer 能夠準確預測蛋白質-配體複合物的結構和構象變化。(資料來源:論文)
預測管線和底層神經網路架構都旨在反映生物分子複合物的多尺度層次結構。具體而言,NeuralPLexer 包括:
(1)基於圖的網絡,將單個小分子和氨基酸圖的原子級化學和幾何特徵編碼為張量表示,透過受物理啟發的網絡架構實現,該網絡架構經過百萬級分子構象和生物活性數據庫的訓練;
(2)接觸預測模組(CPM),在最近的視覺語言模型和折疊預測網絡的推動下,使用基於注意力的網絡生成殘留尺度的分子間距離分佈、粗粒度接觸圖和相關的配對表示;
(3)等變結構去噪模組(ESDM),用於產生以原子尺度和殘留尺度網絡的輸出為條件的結合複雜原子結構,使用等變的結構化去噪擴散過程,並保留蛋白質和配體分子的手性約束。
在對蛋白質-配體盲對接進行評估時,與PDBBind2020 基準上性能最佳的現有方法相比,NeuralPLexer 將預測成功率提高了高達78%。在針對挑戰性目標的配體結合位點設計中,NeuralPLexer 僅使用計算生成的截短支架即可有效恢復高達45% 的結合位點結構。
與現有的基於物理的方法相比,這代表了成功率的質的提高。此外,NeuralPLexer 在選擇性預測受誘導擬合結合或構象選擇影響的蛋白質結構方面比現有方法表現出系統優勢;在兩個具有大結構可塑性的配體結合蛋白基準數據集上,NeuralPLexer 優於最先進的蛋白質結構預測演算法AF2,最高的模板建模得分(TM-score)(平均0.906)以及配體結合後發生重大構象變化的結構域的準確性提高了11-13%。
NeuralPLexer 模擬配體結合和蛋白質結構變化的多功能能力可以快速表徵構象景觀,從而促進更好地理解控制蛋白質功能的分子機制,從而有助於在蛋白質組規模上識別治療幹預和蛋白質工程的非常規靶點。
結語
作為一種數據驅動的方法,NeuralPLexer 具有通用性,並且可以透過整合更好的實驗和生物資訊數據來持續改進。來自更廣泛社區的訓練和基準數據集的管理的改進,可能能夠對沒有實驗確定的同源物蛋白質家族進行更系統的分析,並將該方法擴展到更具挑戰性的系統,例如翻譯後修飾和多態性大型異聚蛋白質複合物。
該研究為探索這些方向提供了通用的計算框架,為快速且準確的蛋白質-配體複合物結構預測鋪平了道路,從而促進結構生物學、藥物發現和蛋白質工程領域的進步。
論文連結:https://www.nature.com/articles/s42256-024-00792-z